植物所等合作完成了粳稻品种中花11端粒到端粒高精度全基因组组装
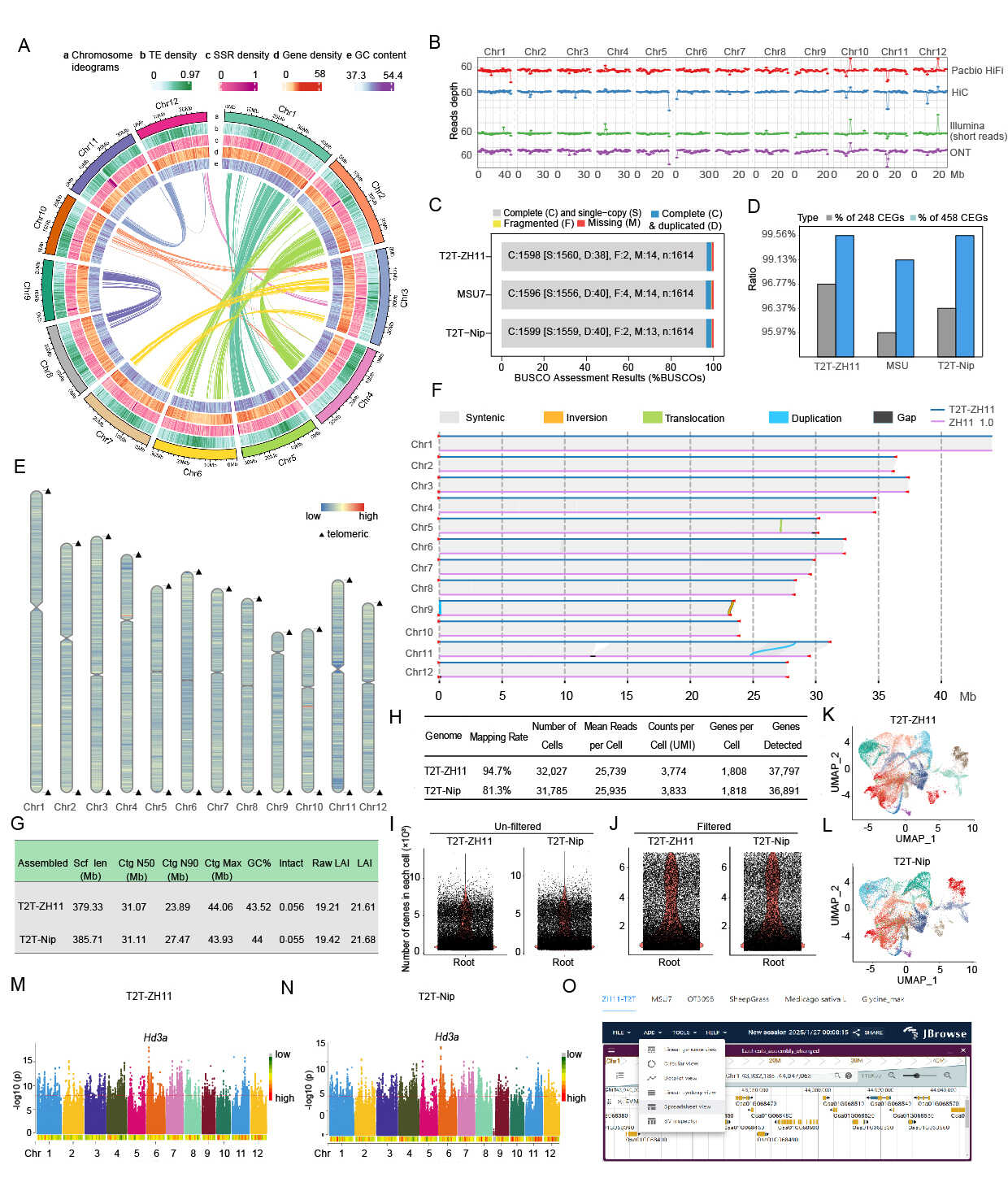
粳稻品种中花11(ZH11)是进行水稻功能基因组研究的模式品种,具有高转化效率、适宜的生育期、良好的物理和化学诱变效率等特点。植物所科研人员等在前人工作的基础上,借助PacBio HiFi(HiFi)和Oxford Nanopore(ONT)长读长测序技术,结合染色体构象捕获、超长DNA光学图谱(Bionano),染色质免疫共沉淀(ChIP-seq)等多组学数据,完成了水稻品种中花11的端粒到端粒(T2T)完整基因组组装。
科研人员累计获得了23 Gb HiFi、56.7 Gb ONT长读基因组片段数据、42.6 Gb染色体构象捕获数据、1.9 Gb全长转录组数据和246 Gb光学图谱、24.96 Gb重测序以及超100 Gb转录组等数据。通过初步组装和进一步筛选优化,最终得到了中花11的端粒到端粒高精度全基因组,其基因组大小为379.33 Mb,包含12条完整染色体,补齐了之前版本的25个缺口,实现了真正意义上的完整组装。新组装的中花11基因组能精准识别出12个着丝粒和24个端粒,科研人员利用ChIP-seq和光学图谱数据进行验证,新增了2.29 Mb新序列,其中包括5号染色体末端和11号染色体的中部关键区域。与前2个版本(ZH11-IGDBv1和ZH11 1.0)相比,科研人员发现了一个新基因Osa05G046820,并通过RNA-seq数据揭示了该基因的表达特征。此外,研究发现9号染色体着丝粒位置与日本晴水稻基因组之间存在显著差异,为染色体进化研究提供了新视角。
该基因组的质量经过了多组学严格验证,通过单细胞转录组、普通转录组、染色质可及性、DNA甲基化等多组学数据比对发现,以T2T-ZH11为参考时,序列比对率、基因检测数量均有提高。在完整性方面,BUSCO评估达99.01%,CEGMA评估达99.56%;碱基质量值QV达49.49,基因组组装质量的评价指数LAI达到“黄金标准”,是当前组装精度和完整度最高的中花11基因组。中花11的端粒到端粒完整基因组组装为水稻功能基因挖掘、分子标记辅助育种、重要农艺性状解析提供了“基因蓝图”,为水稻基础研究和遗传品质改良提供了重要支撑。
中花11完整基因组中共预测到56,948个基因,其中13,028个转座子相关基因,174.07 Mb转座子序列和 18.09 Mb串联重复序列,这些数据为解析水稻基因组进化和表观调控机制提供了重要资源。目前,相关数据已上传至国家基因组数据中心和CROPTILLING(www.croptilling.com/ZH11)等公开数据库。
相关进展于7月22日在线发表于期刊Plant Communications上,植物所助理研究员姚学峰为本文的第一作者,刘春明研究员为通讯作者。中国农业科学院作物科学研究所李慧慧研究员和希望组生物科技有限公司(Grandomics)孙宗毅为本研究提供了支持和指导,北京大学博士研究生许鑫彤参与了此项研究。该研究得到了基金委重点项目、中国农业科学院科技创新工程、国家重点研发计划项目等资助。
原文链接:
https://www.cell.com/plant-communications/fulltext/S2590-3462(25)00225-1
T2T-ZH11参考基因组的组装与验证